https://app.codility.com/programmers/lessons/5-prefix_sums/genomic_range_query/
GenomicRangeQuery coding task - Learn to Code - Codility
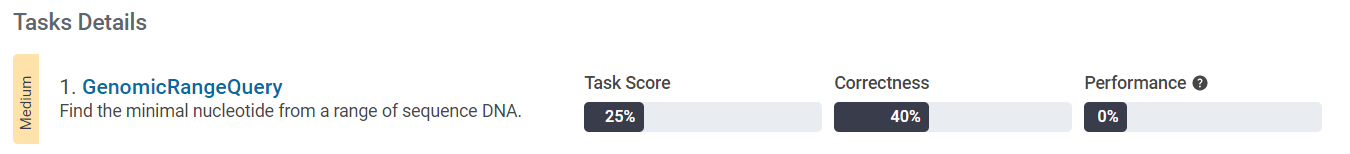
Find the minimal nucleotide from a range of sequence DNA.
app.codility.com
문제
- DNA sequence는 비어있지 않은 배열( S = N characters )이다.
- M queries는 M개의 정수들로 이루어진 P, Q 배열로 비어있지 않은 배열이다.
- A, C, G, T 순서로 1, 2, 3, 4가 매칭되며 M쿼리에 따른 인덱스 범위 안에서 minimal impact factor(제일 낮은 수)의 DNA 서열을 구하라.
- 예시
S = CAGCCTA
M = 3
P = [ 2, 5, 0 ], Q = [ 4, 5, 6 ]
1. 2( P[0] ) ~ 4( Q[0] )사이의 DNA는 'GCC'에서 C = 2, G = 3으로 작은 수는 2
2. 5( P[1] ), 5( Q[1] ) = T = 4
3. 0( P[2] ) ~ 6( Q[2] )는 전범위로 가장 작은 impact factor는 1
-> [ 2, 4, 1 ]
제한조건
- N is an integer within the range [1..100,000];
- M is an integer within the range [1..50,000];
- each element of arrays P and Q is an integer within the range [0..N − 1];
- P[K] ≤ Q[K], where 0 ≤ K < M;
- string S consists only of upper-case English letters A, C, G, T.
N은 1 ~ 100,000사이의 정수
M은 1 ~ 50,000사이의 정수
P, Q의 각각의 원소는 0 ~ N-1사이의 정수
P[K] ≤ Q[K], where 0 ≤ K < M;
S변수는 대문자로 이루어진 A, C, G, T만으로 구성된다.
풀이
- key-value를 활용한 map 자료구조를 활용하여 A,C,G,T에 1,2,3,4를 매칭하여 만든다.
- S는 charArray로 변환하여 활용한다.
- M만큼 for문을 순회하며 P, Q의 인덱스에 해당하는 query들을 풀이한다.
- for문 안에서 조건은 제한조건 P[K] <= Q[K]에 따라서 P[K] == Q[K] or P[K] < Q[K]가 된다.
- P[K] < Q[K]조건에서는 S에 해당하는 인덱스만큼 for문으로 순회하며 최소값을 찾는다.
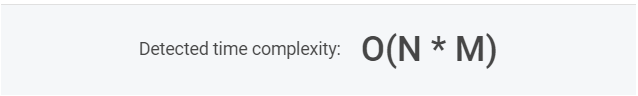
예상 시간복잡도 : ???
소스코드
// you can also use imports, for example:
import java.util.*;
// you can write to stdout for debugging purposes, e.g.
// System.out.println("this is a debug message");
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
// write your code in Java SE 8
Map<Character, Integer> Nucleotide = new HashMap<Character, Integer>(){{
put('A',1);
put('C',2);
put('G',3);
put('T',4);
}};
int[] answer = new int[P.length];
char[] dnaSequence = S.toCharArray();
int M = P.length;
// M만큼 쿼리 반복
for( int i=0; i<M; i++ ){
if( P[i] == Q[i] ){
answer[i] = Nucleotide.get(dnaSequence[P[i]]);
}else{
int min = Nucleotide.get(dnaSequence[P[i]]);
for( int j=P[i]+1; j<Q[i]+1; j++ ){
min = Math.min(Nucleotide.get(dnaSequence[j]), min);
}
answer[i] = min;
}
}
return answer;
}
}

명일 재풀이 해보는 것으로... 머리가 안돌아가는 하루ㅠ
3/27 재풀이
풀이 방법 변경
- 제한조건 P[K] <= Q[K]에 따라서 P[K] == Q[K] or P[K] < Q[K]가 된다.
- P[K] < Q[K]조건에서는 for문 순회가 아닌 배열을 복사( Arrays.copyOfRange )해서 정렬하여 최소값( minimal impact ) 만 찾도록 하고, 같을 경우에는 위의풀이와 동일하게.( 알파벳 순서로 정렬해도 가능 A, C, G, T가 각각 순서대로 이므로 )
// you can also use imports, for example:
import java.util.*;
// you can write to stdout for debugging purposes, e.g.
// System.out.println("this is a debug message");
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
// write your code in Java SE 8
Map<Character, Integer> dna = new HashMap<Character, Integer>(){{
put('A',1);
put('C',2);
put('G',3);
put('T',4);
}};
char[] sArr = S.toCharArray();
int M = P.length;
int[] answer = new int[M];
for( int i=0; i<M; i++ ){
if( P[i] == Q[i] ){
answer[i] = dna.get(sArr[P[i]]);
}else{
char[] tmp = Arrays.copyOfRange(sArr,P[i],Q[i]);
Arrays.sort(tmp);
answer[i] = dna.get(tmp[0]);
}
}
return answer;
}
}
더 안좋은 결과.. 복사해서 정렬하는거 자체가 안좋을 뿐더러, 결과도 제대로 못 찾아내는.. 다시 풀이 방법 부터 다시 생각해보는 것으로...
풀이 방법 변경
- String.substring와 contains로 풀이 하는 방법으로 변경..
- fail...
// you can also use imports, for example:
import java.util.*;
// you can write to stdout for debugging purposes, e.g.
// System.out.println("this is a debug message");
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
// write your code in Java SE 8
Map<Character, Integer> dnamap = new HashMap<Character, Integer>(){{
put('A',1);
put('C',2);
put('G',3);
put('T',4);
}};
int M = P.length;
int[] answer = new int[M];
for( int i=0; i<M; i++ ){
if( P[i] == Q[i] ){
answer[i] = dnamap.get(S.substring(P[i],P[i]+1).charAt(0));
}else{
String tmp = S.substring(P[i],Q[i]+1);
if( tmp.contains("A") ){
answer[i] = 1;
}else if( tmp.contains("C") ){
answer[i] = 2;
}else if( tmp.contains("G") ){
answer[i] = 3;
}else{
answer[i] = 4;
}
}
}
return answer;
}
}
결국 구글링으로 풀이..ㅠ 하위 분의 블로그 참조해서 풀이
https://jackjeong.tistory.com/44
[Codility(코딜리티)] Lesson5. GenomicRangeQuery (100%)
Lesson 5 Prefix Sums - GenomicRangeQuery (Java Solution) app.codility.com/programmers/lessons/5-prefix_sums/genomic_range_query/ GenomicRangeQuery coding task - Learn to Code - Codility Find the min..
jackjeong.tistory.com
풀이
- 각 A,C,G,T에 해당하는 1,2,3,4를 Map에 매칭시킨다.
- countArr( int[][] )와 count( int[] )를 초기화 하고 각
- S를 순차적으로 순회하면서 각 count 배열을 만들어서 매칭시켜놓는다.
예시 : S = CAGCCTA-> [ [0100], [1100], [1110], [1210], [1310], [1311], [2311] ]
P[0] = 2, Q[0] = 4일 때 [1210] - [1100] = [0110] -> for문 순회하며 최초로 0이 아닌 것에서 answer에 저장
- 주의해야 할 사항은 Q[i]의 인덱스는 그대로, P[i]의 인덱스는 1을 빼준다는 점( 개고생을.. )
소스코드
// you can also use imports, for example:
import java.util.*;
// you can write to stdout for debugging purposes, e.g.
// System.out.println("this is a debug message");
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
// write your code in Java SE 8
Map<Character, Integer> dnamap = new HashMap<Character, Integer>(){{
put('A',1);
put('C',2);
put('G',3);
put('T',4);
}};
char[] sArr = S.toCharArray();
int[] counter = {0,0,0,0};
int[][] counterArr = new int[sArr.length][counter.length];
for( int i=0; i<counterArr.length; i++ ){
counter[dnamap.get(sArr[i])-1] += 1;
for( int j=0; j<counter.length; j++ ){
counterArr[i][j] = counter[j];
}
}
int M = P.length;
int[] answer = new int[M];
for( int i=0; i<M; i++ ){
if( P[i] == Q[i] ){
answer[i] = dnamap.get(sArr[P[i]]);
}else{
for( int j=0; j<counter.length; j++ ){
int end = counterArr[Q[i]][j];
int start = P[i] == 0?0:counterArr[P[i]-1][j];
if( end - start > 0 ){
answer[i] = j+1;
break;
}
}
}
}
return answer;
}
}


이 한문제에 몇시간을 투자하는건지.. 꾸준히 해야겠다 ^^;;
'코딜리티 문제풀이' 카테고리의 다른 글
| Codility Lesson 06 - Distinct( JAVA ) (0) | 2022.03.28 |
|---|---|
| Codility Lesson 05 - MinAvgTwoSlice( JAVA ) (0) | 2022.03.28 |
| Codility Lesson 05 - CountDiv( JAVA ) (0) | 2022.03.21 |
| Codility Lesson 05 - PassingCars( JAVA ) (0) | 2022.03.20 |
| Codility Lesson 04 - MissingInteger( JAVA ) (0) | 2022.03.20 |



